Présentation
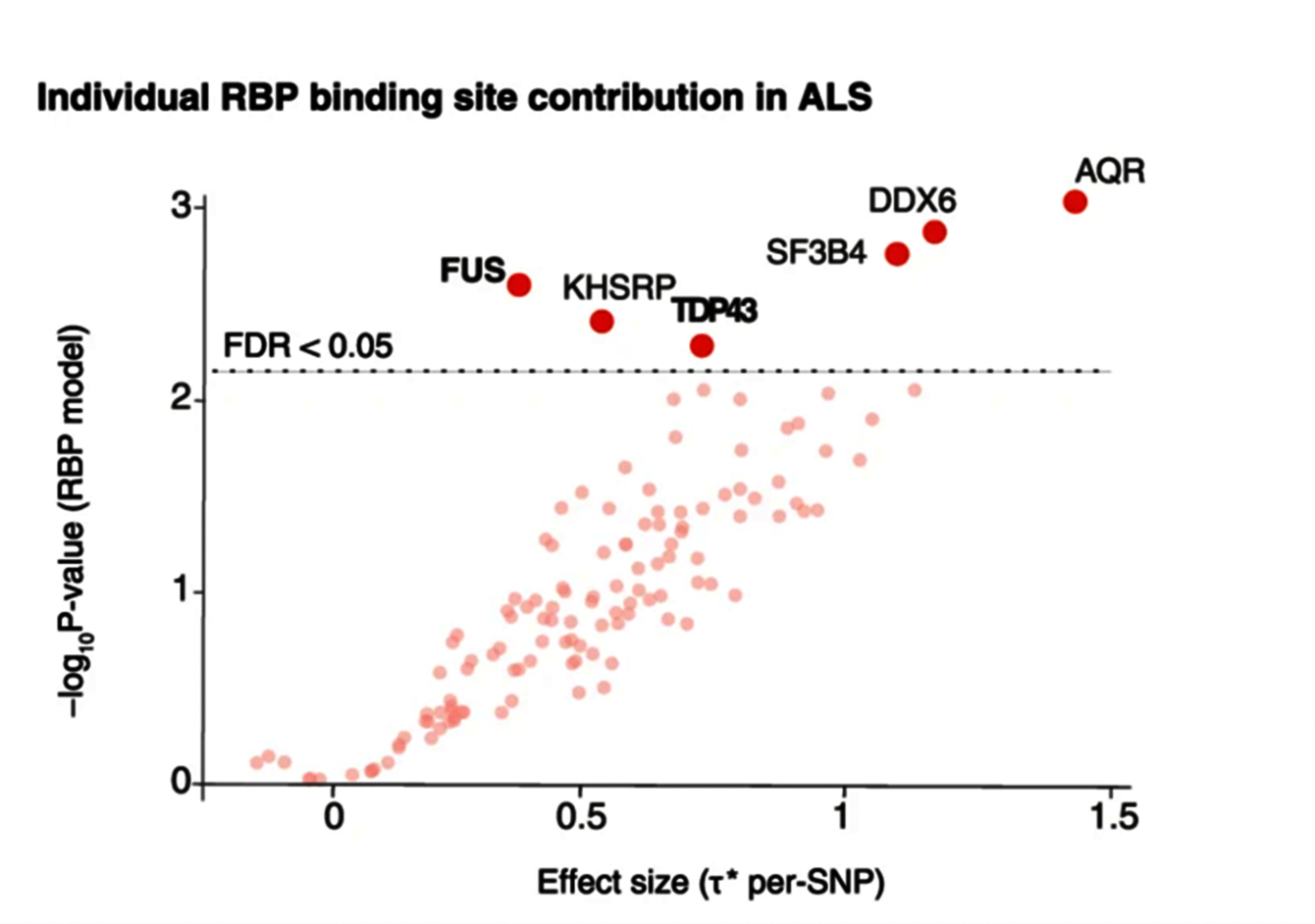
Graphique montrant l'héritabilité de la SLA significativement enrichie dans les sous-types de sites de liaison des protéines de liaison à l'ARN tels que FUS et TDP43. (Megat et al., 2023)
La sclérose latérale amyotrophique (SLA) est une maladie neurodégénérative mortelle qui se manifeste par une faiblesse musculaire et une paralysie progressives, entraînant la mort dans les deux à trois ans suivant l'apparition des symptômes. La SLA résulte de la dégénérescence progressive de deux populations neuronales impliquées dans le contrôle volontaire de la motricité : les neurones corticospinaux glutamatergiques (CSN, ou motoneurones supérieurs) dans le cortex moteur, et les motoneurones cholinergiques (MN, ou motoneurones inférieurs), dans le tronc cérébral et la moelle épinière. Les gènes responsables ont été identifiés dans plus de la moitié de ces cas, les plus représentés étant la sous-unité du complexe C9orf72-SMCR8 (C9orf72), la superoxyde dismutase 1 (SOD1), la protéine de liaison à l'ADN TAR (TARDBP) et la protéine de liaison à l'ARN FUS (FUS). Bien que la génétique de la SLA soit complexe, les mutations causales convergent remarquablement vers des voies dysrégulées récurrentes, notamment la réparation de l'ADN, le métabolisme de l'ARN et des protéines, le trafic intracellulaire et le dysfonctionnement mitochondrial.
Dans notre équipe, nous utilisons l'intégration de données multi-omiques en utilisant l'ARNseq de noyaux uniques de neurones vulnérables et résistants ainsi que des ensembles de données génétiques à grande échelle pour découvrir de nouveaux facteurs de risque et de protection associés à la SLA et la DFT. En utilisant des motoneurones dérivés de cellules souches pluripotentes induites, nous visons à valider nos nouveaux gènes cibles et à mieux comprendre les mécanismes qui sous-tendent la neuroprotection ou la neurodégénérescence dans la SLA.







